Ein Beitrag von Sahil Sharma und Cynthia Sharma auf der Konferenz „CRISPR 2023“ in Würzburg.
CRISPR-Cas dient bei Bakterien in erster Linie zur Verteidigung gegen Virusinfektionen. Aber ist das alles? Sahil Sharma und Cynthia Sharma von der Universität Würzburg haben in ihrem Forschungsobjekt, dem Lebensmittelkeim Campylobacter jejuni genauer hingeschaut, und finden tatsächlich weitere, zusätzliche Funktionen.
Zur Erinnerung
Bei einer Virusinfektion können Bakterien DNA-Schnipsel des Virus an eine ganz bestimmte Stelle (CRISPR-Array) in ihr Genom einbauen. Die Schnipsel werden in RNA-Moleküle überschrieben (crRNA) und die dient als „Adressaufkleber“ um die „Genschere“, das Cas-Enzym zur Virus-DNA zu leiten und diese zu zerschneiden. Damit wir die Ausbreitung des Virus verhindert.
Ganz so einfach ist es aber nicht! In einer Bakterienzelle schwimmen Unmengen an RNAs mit anderen wichtigen Funktionen herum – die dürfen nicht versehentlich als „Adressaufkleber“ verwendet werden! crRNAs müssen deshalb eine Art „Signal“ enthalten, um von dem Cas-Enzym eindeutig erkannt zu werden.
Das sieht so aus:
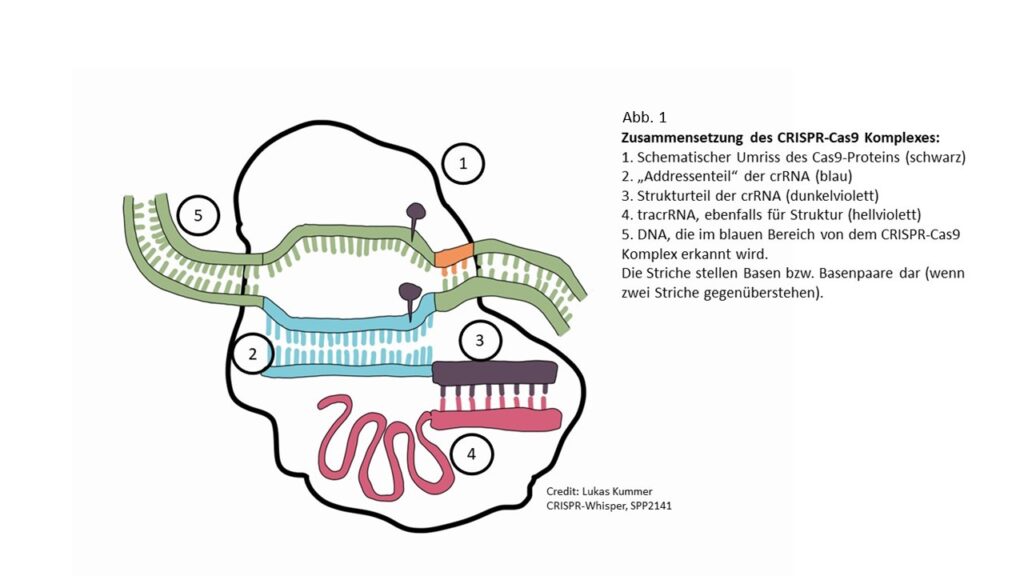
An der eigentlichen Adresse der crRNA hängt noch ein Stückchen dran, das mit einer bestimmten Faltung das Cas-Enzym erkennt. Dazu kommt noch eine zweite RNA (tracrRNA), die die Erkennung perfekt macht.
So weit, so gut.
Wie man mehr herausfindet
Mit einer pfiffigen Methode (RIP-Seq) die wir an anderer Stelle erklärt haben, kann man aus Bakterien alle RNAs herausziehen, die an das Cas-Enzym gebunden sind. Die RNAs werden alle sequenziert, sortiert und gezählt.
Wie erwartet findet man sehr viel von der tracrRNA und die verschiedenen crRNA Schnipsel.
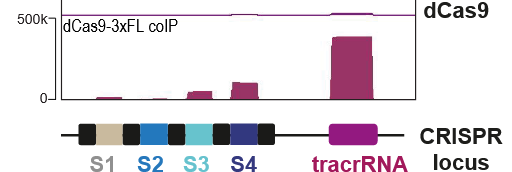
Abb. 2:
In der Abbildung ist unten die Anordnung im CRISPR-Locus auf der DNA gezeigt. Die farbigen Boxen S1 bis S4 geben vier „Adressaufkleber“(Spacer) an, Abschnitte der DNA, die in crRNA überschrieben werden. Dazwischen liegen (schwarz) die „Repeats“ die alle gleich sind und die „Anhängsel“ an die crRNA bilden. Das sind die speziell gefalteten Abschnitte, die durch ihre Struktur das Cas-Enzym erkennen. Rechts davon kommt das Gen für die tracrRNA.
In der Box darüber sind sehr komprimiert die Ergebnisse der RNA-Zählung gezeigt. Die violetten Balken bestehen eigentlich aus sehr vielen Zeilen, die die Sequenz der RNA angeben. Die Zeilen sind über der DNA Sequenz auf der unteren Karte gestapelt. Links neben der Box ist angegeben, dass die volle Höhe 500.000 Zeilen, d.h. gefundene RNAs für einen Ort entspricht. In diesem Experiment hat man also etwa 400.000 tracrRNA und etwa 100.000 RNAs von S4 gefunden. (Warum S1 und S2 so deutlich weniger RNAs haben, ist eine andere Geschichte!)
Wenn man genauer hinschaut, findet man aber mehr! Besonders gut, wenn man den Hintergrund der normalen crRNAs und tracrRNAs entfernt, indem man den entsprechenden DNA Abschnitt des CRISPR-Lokus in den Bakterien ausschneidet. Das Cas-Enzym wird zwar noch produziert, es gibt aber keine crRNAs und tracrRNAs mehr (untere Zeile in Abb. 3).
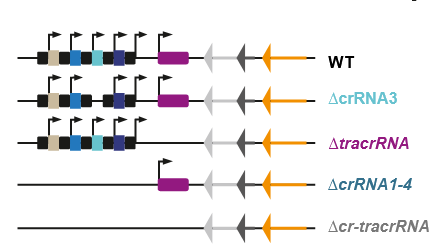
Abb. 3
Genomkarten des CRISPR-Locus in C. jejuni und gentechnisch hergestellten Varianten. Oben ist die Anordnung in den normalen Bakterien, dem „Wildtyp“ gezeigt: links die vier crRNA Blöcke, dann das Gen für die tracrRNA. Rechts sind durch die Pfeilspitzen drei Gene für verschiedene Cas-Enzyme symbolisiert.
In der zweiten Zeile wurde der Ort für Spacer S3 entfernt, in der dritten Zeile die tracrRNA, in der vierten alle Spacer und in der fünften alle Spacer und die tracrRNA. Die Gene für die Cas-Enzyme bleiben erhalten.
Dafür werden jetzt andere RNAs, die an die Genschere binden, sichtbar, die überhaupt nichts mit dem CRISPR-Lokus zu tun haben! Das sind nicht viele, aber zu viele um sie als zufälliges Hintergrundrauschen abzutun. Und es gibt mehr davon, wenn die eigentlichen crRNAs und die tracrRNA ausgeschaltet werden.
Weil die komplette DNA Sequenz des Bakteriums bekannt ist, findet man relativ einfach heraus, zu welchem Gen der Schnipsel passt.
Was da genau passiert, weiß man (noch) nicht. Die mRNA des Gens könnte zerschnitten werden oder der Cas-Komplex sitzt dort einfach und verhindert die Übersetzung in das entsprechende Protein.
Was hat das mit der Wirklichkeit zu tun?
Sagen solche Ergebnisse etwas über die Wirklichkeit aus, oder machen Bakterien, die man gentechnisch so „malträtiert“ hat einfach nur „komische Sachen“? Viele Experimente an Bakterien, Pflanzen und Tieren zeigen, dass die Daten schon weitgehend die Realität wiedergeben. Trotzdem ist die Frage berechtigt. Von Campylobacter jejuni wurde in einem Patienten zufällig ein Stamm gefunden, eine natürliche Variante, die zwar die Gene für das Cas-Enzym und die tracrRNA hat, aber keinen CRISPR-Array und deshalb auch keine crRNAs produziert. Das entspricht fast genau einer der konstruierten Varianten (vierten Zeile in Abb. 3). Und in diesem Stamm bindet Cas9 ebenfalls an die ungewöhnliche RNA – und damit sind wir mitten in der Wirklichkeit!
Es sieht also so aus, dass das Cas-Enzym auch ohne tracrRNA und ohne „richtige“ crRNAs in die eigene Genregulation der Bakterienzellen eingreifen kann. Wie das genau funktioniert und welche Bedeutung das gerade bei pathogenen Bakterien hat, gilt es zu erforschen.
Es gibt noch viel zu tun – aber es ist inzwischen klar, dass CRISPR-Cas mehr kann, als nur die Verteidigung gegen Viren! Das kennen wir auch schon aus einem ganz anderen CRISPT-Cas System, das Selina Rust und Meral Kara hier beschrieben haben.
Und was bringt das jetzt?
Grundlagenforschung ist zunächst „Neugierforschung“ weil man verstehen will, wie Leben funktioniert. Bei CRISPR-Cas kommt hinzu, dass das System bereits in vielen Bereichen angewendet wird (Medizin, Pflanzenzucht, Materialforschung u.a.). Deshalb muss man möglichst viel darüber wissen. (Nein, man kann niemals „alles“ wissen, bevor man in die Anwendung geht!). Was Cynthia und Sahil hier erforscht haben, ist von der Anwendung gar nicht weit entfernt! Zusammen mit ihrem Kollegen Chase Beisel beschreiben sie im „Laborjournal“, wie sie aus ihren Ergebnissen ein Werkzeug für die medizinische Diagnostik entwickeln.
Dieser Artikel ist etwas schwieriger zu lesen, für Laien aber noch immer verständlich.
Vielen Dank an Cynthia und Sahil, für ihre Daten und die Unterstützung bei diesem Beitrag!
Author
W. Nellen, BioWissKomm
Titelbild:
Sheree Domingo für Science Bridge