Leitung: Franz Baumdicker
Mitarbeitende: Klara Burger, offene Stellen zu vergeben (siehe letzter Abschnitt)
Standort: Universität Tübingen
Kurzbeschreibung des Projekts:
Franz Baumdicker beschäftigt sich mit mathematischen Modellen, mit denen die Evolution und Ökologie der Gene und Genome von Mikroorganismen beschrieben werden soll. Hier geht es nicht um experimentelle Arbeiten im Labor sondern hauptsächlich um Mathematik und Bioinformatik. Dabei werden Daten und Ergebnisse aus der experimentellen Forschung, z.B. die anderer Projekte aus dem Schwerpunktprogramm, eingesetzt.

Die revolutionäre CRISPR-Cas-Technologie zur präzisen Veränderung von Genomen hat ihren Ursprung in den natürlichen CRISPR-Systemen in Bakterien und Archaea. Viele dieser Systeme funktionieren als Abwehrsysteme gegen die Infektion durch Viren. Darüber hinaus können CRISPR-Systeme die Evolution von Bakterien jedoch auch direkt beeinflussen, ohne dass Viren beteiligt sind.
Ziel dieses Projektes ist die Entwicklung und Anwendung neuer mathematischer Modelle für die Evolution von CRISPR in Prokaryoten. Viele wissenschaftliche Arbeiten haben sich bereits mit dem Zusammenspiel zwischen Bakterien mit CRISPR und den viralen Parasiten beschäftigt. Im Gegensatz dazu konzentrieren wir uns auf die Mechanismen der Evolution von CRISPR-Cas-Systemen in Bakterien und Archaea, die ohne virale Einflüsse beschrieben werden können. Insbesondere möchten wir folgende Fragen beantworten:
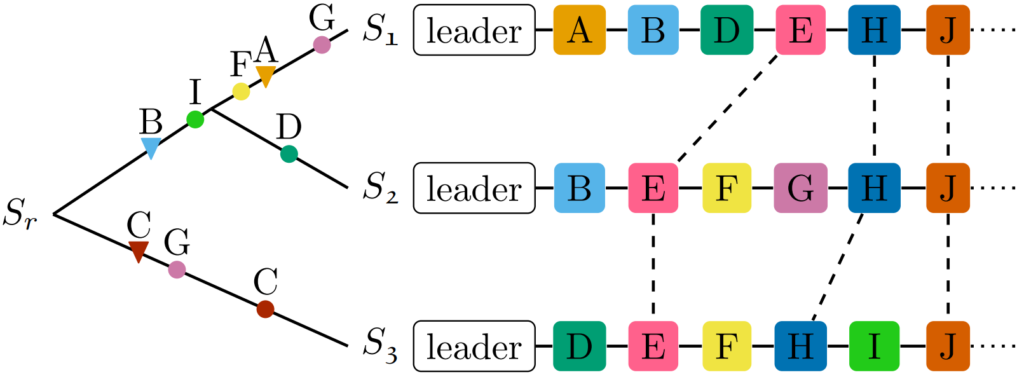
• Die CRISPR-Systeme enthalten kurze, sich immer wieder wiederholende identische DNA-Sequenzen, die von den sogenannten “Spacern” (Abstandhalter) unterbrochen werden. Diese stammen meistens von eingedrungenen Viren ab und liefern das “Adress-Label” für die Cas-“Genschere”.
Weil die Spacer in chronologischer Reihenfolge früherer Infektionen angeordnet sind, bieten sie einen einzigartigen Einblick in die Evolutionsgeschichte (d.h. die “Krankheitsgeschichte”) der Bakterien. Können wir die beobachteten Muster innerhalb der Spacer-Abfolge erklären und das Vorkommen von Spacern unter bestimmten Selektionsbedingungen beobachten? Können wir rekonstruieren, unter welchen Bedingungen ein Spacer in der Vergangenheit aufgenommen wurde, um die Evolution von CRISPR-Cas in Bakterien besser zu verstehen?
• Wenn versehentlich eigene Gensequenzen als Zielsequenz für CRISPR benutzt werden, führt dies normalerweise zum Tod des Bakteriums. Können wir die Wirkung von gegen das Bakterium selbst gerichteten (selbst-targetierenden) CRISPR-Systemen aus den Genomsequenzen von Bakterien nachvollziehen oder sogar modellieren?
• Es gibt CRISPR-Systeme, deren Spacer gegen Genvarianten in “Verwandten” derselben Population gerichtet sind. Bei einem genetischen Austausch (Rekombination) mit diesen Verwandten verhindert dies wahrscheinlich den Austausch oder führt eventuell sogar zum Tod des Empfängers. Damit wird der Austausch und die Verteilung von Genen innerhalb einer Population beeinflusst.
Der Genaustausch mit den betroffenen “Verwandten” kann erst dann wieder stattfinden wenn die Ziel-Gene verändert oder, wenn möglich, ganz verloren wurden. Wir wollen verstehen, wie sich CRISPR-Arrays und ihre Ziel-Gene im eigenen Genom und im Genom von Verwandten dynamisch verändern.
Bisher wurde dieses Projekt von der DFG als sogenannte „Eigene Stelle“ finanziert. Inzwischen hat Franz Baumdicker eine unabhängige Forschungsgruppe „Mathematical and Computational Population Genetics“ im Exzellenzcluster „Controlling Microbes to Fight Infections“ in Tübingen gestartet und sucht nach motivierten Doktorand*innen die sich für die Evolution von CRISPR interessieren. Die Stellenausschreibung kann unter folgendem Link gefunden werden:
http://www.baumdickergroup.de/index.php/join-us?id=21:phd-position-crispr&catid=2
